


サル/ヒト免疫不全ウイルス(SHIV)は後天性免疫不全症候群(AIDS)の病態解明を探索する重要なツールとして利用されています。SHIVには異なる病原性を示す複数の株が存在しますが、それらの定量的な感染動態についてはほとんど研究が進んでいませんでした。数理生物学研究室の岩波翔也さんらの研究グループは、数理モデルを用いて培養細胞から得られたウイルス感染実験データを解析し、感染性のあるウイルスの産生効率の違いが、高病原性と弱病原性を決定付ける要因のひとつであることを明らかにしました。研究成果はTheoretical Biology and Medical Modellingに発表されました。
後天性免疫不全症候群(AIDS)は、ヒト免疫不全ウイルス(HIV)が免疫細胞に感染し、免疫細胞を破壊して後天的に免疫不全を起こす疾患です。AIDSの医療研究が難しいと言われる理由のひとつとして、HIVがヒトとチンパンジーにしか感染しないために動物実験が難しいことが挙げられます。これを克服するため開発されたのが、HIV-1とサル免疫不全ウイルス(SIV)の特徴をあわせもつキメラウイルス[1]であるサル/ヒト免疫不全ウイルス(SHIV)です。SHIVは代表的な実験動物であるアカゲザルに感染するため、AIDS/HIV医療の発展に大きく役立つことが期待されています。
SHIVには多くのウイルス株[2]が存在し、株ごとに異なる病原性[3]を示します。この病原性の違いがウイルス感染動態の何によるものなのかはこれまでわかっていませんでした。岩波さんらは、病原性が異なる2種類の株を用いた培養細胞での感染実験データを、数理モデルにより解析し、病原性の違いの原因を解明しました。
SHIVウイルスはサルに感染しますが、より詳しく言うとサルの免疫細胞に感染します。図1は、細胞レベルで何が起こっているのかを描いたものです。感染していない細胞(標的細胞)は、ウイルスが感染することで感染細胞となります。感染細胞はウイルス粒子を放出します。放出されるウイルスには2種類あり、ひとつは新たな標的細胞に感染できる感染性ウイルス、もうひとつは感染性を持たない非感染性ウイルスです。さらに感染性ウイルスは時間がたつと感染性を失って、非感染性ウイルスへと変わっていきます。特に重要なのは感染性ウイルスの数で、生体内で感染性ウイルスが効率よく増えていけば標的の免疫細胞の数が減っていき、AIDSが発症してしまいます。反対に、感染性ウイルスの数が減り続けてゼロに近づけば発症には至りません。
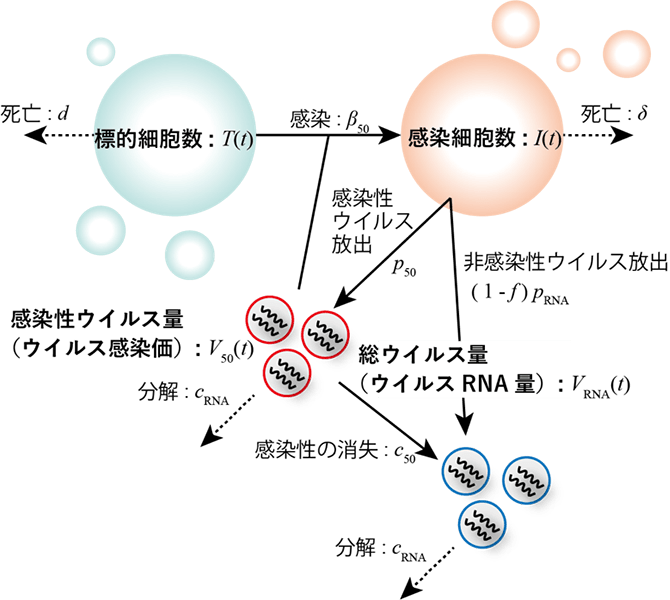

以上の細胞レベルの視点では、病原性が高くなる、つまり感染性ウイルスが増えやすい理由として
など様々なものが考えられます。今回の研究では、実際の理由がこのうちのどれなのか(1つとは限らない)に迫りました。
実験班(世界で初めてSHIVの開発に成功した京都大学ウイルス研究所・三浦智行准教授らのグループ)は、高病原性のSHIV-KS661と低病原性のSHIV-#64を培養細胞に感染させ、9日間の標的細胞数、感染細胞数、総ウイルス量、感染性ウイルス量を計測しました。
これらの計測されたデータは、いわば結果です。そして今回知りたいのは原因です。このように結果から原因を推測するとき、数理モデルが役立ちます。岩波さんたち数理モデリング班は図1に描かれた細胞とウイルスのふるまいを元に、標的細胞数、感染細胞数、総ウイルス量、感染性ウイルス量の時間変化を連立微分方程式であらわしました(図2)。
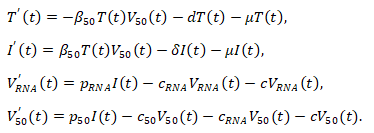
図2の連立方程式は高病原性株と低病原性株のどちらにもあてはまりますが、式に含まれているパラメータ(β50, pRNA, p50, δ, cRNA)の値には株ごとの特徴があると考えられます。この数理モデルを用いて、培養細胞でのウイルス感染実験データを解析し、高病原性株と低病原性株のパラメータを推定しました(図3)。
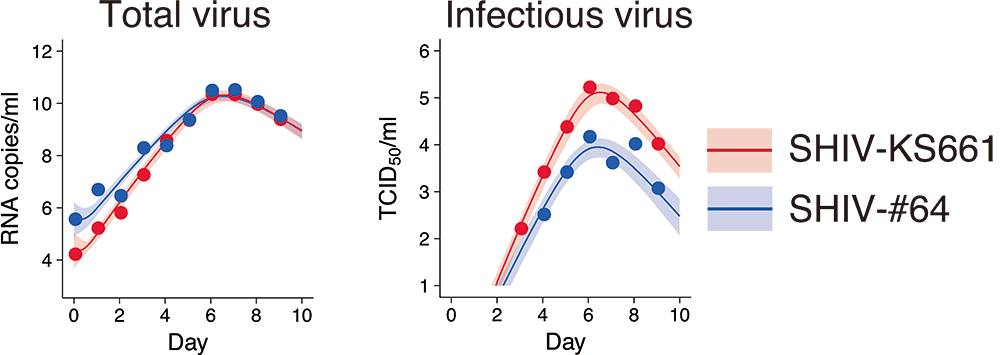

推定したパラメータを用いて、ウイルスの感染性の指標となるマルサス係数(ウイルス感染初期の増殖速度)と基本再生産数(ひとつの感染細胞から感染する二次感染細胞数)を計算しました。マルサス係数と基本再生産数はそれぞれ、ウイルスの適応度(増殖の有利さ)と感染力に対応します。これらの値を高病原性株と低病原性株で比較すると、どちらも高病原性株の方が大きいことがわかりました。高病原性株は個体レベルの視点ではサルに免疫不全を引き起こす株であり、体内の細胞レベルの視点ではサルの体内で広がりやすい株であると言えますが、そのことがシャーレ上での細胞実験からも確認されました。
さらに数理モデルを用いて、高病原性株の適応度と感染力が低病原性株よりも高い原因を知るために、バーストサイズ(ひとつの感染細胞が放出するウイルス量)を計算・比較しました。すべてのウイルス(感染性+非感染性)についてのバーストサイズは、高病原性株と低病原性株とで差がありませんでした。先ほど病原性の違いにつながる理由として考えられるものを4例あげましたが、そのうち「(2) ひとつの感染細胞が放出するウイルスの総量が多い」ではなかったということになります。
一方、感染性ウイルスについてのバーストサイズは高病原性株の方が大きいことがわかりました。実際に、培養細胞での感染実験中の総ウイルス量に対する感染性ウイルス量を比較すると高病原性株では低病原性株に比べて10倍近い差があることがわかりました。このことから、高病原性株は低病原性株に比べて、感染性ウイルスを効率的に産生するといえます。つまり、「(3) 放出されるウイルスのうち、感染性ウイルスの割合が高い」が病原性の違いをもたらす原因のひとつだったのです。
今回は培養細胞でのウイルス感染実験データを、数理モデルを用いて解析することで、SHIVの病原性の違いが感染性ウイルス粒子の生産効率の違いで説明できる可能性を定量的に説明することに成功しました。今後、アカゲザルにおける感染実験データの解析を行い、AIDSの病態解明につなげていく予定です。
本研究で、ウイルス感染実験データの解析から感染性ウイルス産生効率を定量化したように、数理モデルを用いた生物学の研究では、実験だけでは見ることのできない生物学的側面を知ることができます。 「生物なのに数式??」と思われるかもしれませんが、実験科学と数理科学が融合することで、生物学(そして医学)がさらに発展することが期待されます。
「生物なのに数式??」と思われるかもしれませんが、実験科学と数理科学が融合することで、生物学(そして医学)がさらに発展することが期待されます。
Note:
より詳しく知りたい方は・・・